In het jaar 2000 werd aangekondigd dat het volledige humane DNA bepaald was. Maar was dat wel zo? De nieuwste biotechnologie en bioinformatica technieken zetten twintig jaar later de gebrekken van het meest ambitieuze project in de biologie recht.
In het jaar 2000 werd door de Amerikaanse president Bill Clinton aangekondigd dat wetenschappers voor het eerst de volledige DNA code van de mens hadden bepaald. Alle stukken DNA van een levend wezen samen wordt het genoom genoemd, en dit ambitieuze project was het Human Genome Project. Dit werk vereiste tien jaar van internationale samenwerking en een totale kostprijs van 5 miljard dollar (gecorrigeerd voor inflatie), en het was het helemaal waard. Voor de biomedische wetenschappen had dit project een enorme impact.
Bij het samensmelten van de eicel en de zaadcel komt er DNA van onze ouders samen om zo een nieuwe, unieke combinatie te maken die er nog nooit is geweest, en er na ons ook nooit meer zal zijn. Zelfs het DNA van identieke tweelingen is niet helemaal hetzelfde. Doordat we een set DNA krijgen van beide ouders hebben we bijna alles dubbel, met als uitzondering dat de meeste mannen 1 X en 1 Y chromosoom hebben en we het DNA in de mitochondriën, de energiefabriekjes van de cel, enkel van onze moeder krijgen.
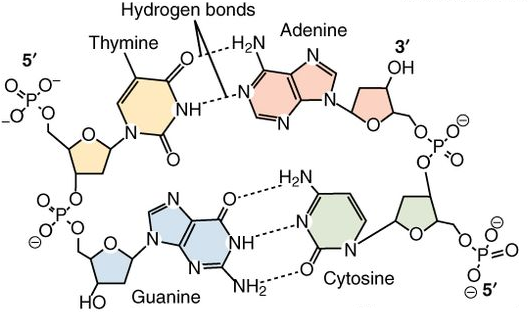
Het DNA zijn lange slierten van vier biochemische 'letters': de moleculen adenine (A), thymine (T), guanine (G) en cytosine (C). Het menselijk genoom is in totaal 2x 3 miljard DNA letters en bevat ongeveer 2x 44000 genen. Een gen is een stuk DNA dat door de cel wordt overgeschreven in een RNA molecule. RNA lijkt wel wat op DNA, maar het DNA blijft veilig in de celkern opgeslagen terwijl het RNA het werk voor het onderhoud van de cel gaat uitvoeren. Bij 20000 van onze genen bevat dat RNA de instructies voor het maken van een eiwit, de rest doet zijn job gewoon als RNA. Alles wat een cel of orgaan maakt of doet ligt ergens opgeslagen in die 6 miljard A, T, G of C moleculen, en tenzij er iets grondig mis gaat dan vormt dat een volledige mens.
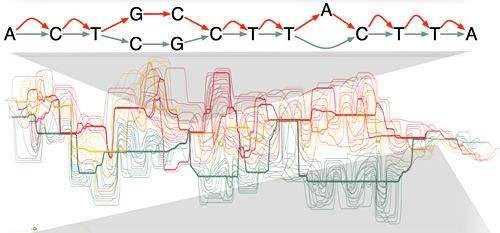
Die 44000 genen nemen samen slechts 5% in van het volledige DNA. Voor de rest zijn er ook stukken die belangrijk zijn voor wanneer, hoeveel en wat voor RNA er juist geproduceerd wordt. En dan zijn er nog stukken waarvan we niet weten wat hun nut is, en waarvan het ook niet zeker is dat ze wel een functie hebben. Misschien liften ze wel gewoon met de rest van ons DNA mee. Het proces van het bepalen van de volgorde van de DNA letters heet sequencing, en daar komt heel wat straffe biotechnologie bij kijken. Na het sequencen moeten de gelezen DNA fragmenten aan elkaar gepuzzeld worden met bioinformatica software, en dit proces heet assembleren. Voor het menselijke genoom werd dat sequencen en assembleren een wedstrijd, want zowel een internationale samenwerking van universiteiten als het bedrijf Celera hadden interesse. Uiteindelijk waren beide zowat tegelijk klaar.
Onvolledig en niet helemaal juist
Maar hadden ze bij het afronden van het project wel het volledige DNA bepaald? Nee, eigenlijk niet. De echt moeilijke stukken, samen zo'n 10%, waren grotendeels nog niet gelukt. Dit zijn vooral stukken waarin eenzelfde DNA fragment heel vaak achter elkaar herhaald wordt, waardoor de puzzel bijna onmogelijk juist te leggen valt tijdens het assembleren. Bijvoorbeeld in het midden van elk chromosoom, het centromeer, zit een stuk van miljoenen letters waarin een specifiek fragment van 170 letters heel vaak bijna identiek herhaald wordt. Ook aan de uiteinden van de chromosomen, wat de telomeer genoemd wordt, zitten vervelende stukken DNA. Ook problematisch zijn lange DNA stukken (minstens 10000 letters, vaak miljoenen) die op verschillende plaatsen voorkomen verspreid in het genoom. Dit heet een segmentele duplicatie. Hierbij kan men niet goed achterhalen waar een bepaald fragment nu juist hoort, of hoeveel keer het nu juist bestaat. Gelukkig zijn er in die stukken relatief weinig genen te vinden, maar ze zijn daarom niet onbelangrijk. Meer nog, hier kunnen vaak nieuwe genen ontstaan, waarvan sommige uniek zijn voor mensen. Misschien ligt de genetische informatie die ons zo'n uitzonderlijke hersenen geeft ergens in de meest vervelende regio's van het genoom verstopt.
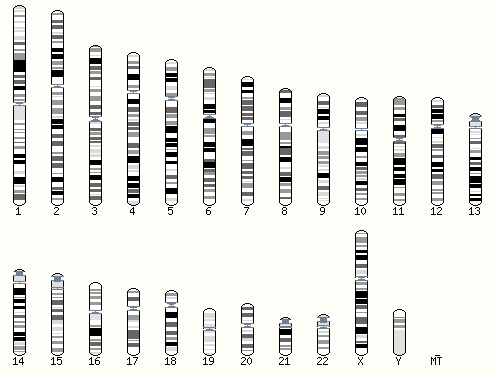
We wisten door microscopisch onderzoek al lang dat het menselijk DNA bestaat uit 23 paren chromosomen in de celkern (23 van de eicel van je moeder en 23 van de zaadcel van je vader). Het stuk DNA in de mitochondriën is heel kort en niet erg complex en dus in deze context niet zo belangrijk. Met de microscoop konden ook al grote genetische fouten opgespoord worden, zoals een extra chromosoom 21 in het syndroom van Down of het uitwisselen van een stuk DNA tussen chromosoom 9 en 22, wat tot leukemie kan leiden.
Maar het was tijdens het assembleren niet gelukt om de volledige puzzel te laten kloppen. In het DNA bleven wel 150 000 gaten over, waar we niet juist wisten hoe de stukken aan elkaar pasten. Vaak wisten we wel dat fragment B achter fragment A kwam, maar konden we niet ontcijferen wat het DNA tussen deze stukken juist was. Er werd ook gemiddelde elke duizend DNA letters wel een verkeerde letter genoteerd in deze eerste versie, wat toch behoorlijk veel fouten zijn. Er werd ook DNA gebruikt van meerdere personen, waardoor het genoom een mengeling werd. Ook was het vaak niet mogelijk om het verschil goed te zien tussen de chromosoom gekregen van de moeder en van de vader. En waar er dan verschillen waren tussen die twee, werd er willekeurig eentje gekozen of fouten gemaakt. Tenslotte zijn er ook plaatsen in dat eerste menselijke genoom waar een heel zeldzame variant voorkomt, en zowat iedereen die je tegenkomt heeft op die plaats een andere DNA letter. Ook dat zorgt voor problemen.
Dus het menselijke genoom was niet volledig en niet correct. Sinds de eerste publicatie werd met behulp van steeds betere biotechnologie nog geregeld een nieuwe versie uitgebracht, waarin foutjes gecorrigeerd werden en gaten opgevuld. In huidige versie, uit 2013, zijn nog (slechts) 350 gaten. Maar ondanks al die gebreken van dat menselijk genoom is de biologie en biomedisch wetenschap compleet veranderd. Ook voor vele andere diersoorten, planten, bacteriën en virussen werden genomen samengesteld. Voortaan kan dat het genoom van een soort gebruikt worden als referentiepunt (het referentiegenoom) waarmee alle studies hun resultaten kunnen vergelijken. Op deze manier moet voor elk nieuw individu niet opnieuw het genoom geassembleerd worden.
Betere biotechnologie
De afgelopen jaren werd voor honderdduizenden personen het DNA, of stukjes daarvan, bepaald en vergeleken met de referentie. Sinds 2007 gaat dat steeds sneller en goedkoper, met een volgende generatie aan sequencing technologieën. Hierdoor zijn heel veel genetische varianten of mutaties gevonden die belangrijk bleken voor menselijke eigenschappen, zoals lichaamslengte, maar ook voor ziektes, zoals de ziekte van Alzheimer, progeria of doofheid. Dit leidt tot een betere diagnose, en meer inzicht naar wat er juist gebeurt bij zo'n aandoening. Als we zo de ziekte beter begrijpen kan dat weer gaan bijdragen tot het vinden van een therapie.
De biotechnologie heeft de laatste jaren ook niet stil gezeten, en er zijn nu methodes om veel langere DNA stukken in één keer te lezen, waardoor de puzzel wel heel wat eenvoudiger wordt. Met de langere sequencing technologieën zijn ook de heel repetitieve stukken van het DNA niet meer zo'n onoverkomelijk probleem. Recent heeft een samenwerking van verschillende onderzoeksinstituten voor de eerste keer het volledig DNA van de mens in elkaar gepuzzeld, in één stuk voor bijna elke chromosoom. Ze noemen zichzelf het telomere-to-telomere consortium en hebben als doel om het DNA in één stuk te assembleren van de ene telomeer naar de andere. Dit huzarenstukje maakt gebruik van de nieuwste technologieën op het vlak van sequencing en specifiek hiervoor ontworpen software, gevolgd door manueel de laatste puzzelstukjes op zijn plaats te leggen. De onderzoekers moeten nog een laatste stuk repetitief DNA ontwarren, op het korte einde van chromosomen 13, 14, 15, 21 en 22, en dan is het eerste volledige genoom een feit. Wellicht krijgt dit veel minder aandacht dan in het jaar 2000, maar het is en blijft een mijlpaal.
Bij dit project werd wel een handig trucje toegepast. Als bron van het DNA werd CHM13 gebruikt, laboratorium cellen waarbij door een biologische fout na de bevruchting twee bijna identieke kopieën van de chromosomen van de vader aanwezig zijn en geen DNA meer van de moeder. Dit wordt een 'hydatidiform mole' genoemd en is niet levensvatbaar. Aangezien er geen DNA van de moeder is wordt de puzzel weer een stukje eenvoudiger, omdat er tussen de chromosomen onderling geen variatie meer is. Dus nu, twintig jaar later, hebben we eindelijk een volledig humaan genoom.

Menselijke diversiteit
Maar het grootste probleem dat overblijft is dat men eigenlijk ook maar één menselijk genoom in elkaar gepuzzeld. Het DNA is bij alle mensen een beetje anders en er bestaat niet zoiets als het menselijke genoom. Het genoom van twee willekeurige personen is voor zo'n 99.4% hetzelfde, maar in die 0.6% zitten wel zo'n 3 miljoen kleine verschillen (van 1 of enkele DNA letters) en 25 000 grote verschillen. Deze grote verschillen zijn meestal veranderingen tussen de 50 - 500 letters, maar soms ook miljoenen DNA letters. Hoe bestudeer je een stuk DNA van een persoon als het referentiegenoom dat stuk niet heeft of heel anders is op die locatie?
Om ook op dat probleem een antwoord te bieden zal de komende jaren ook nog perfecte genomen worden samengesteld van enkele honderden mensen van diverse afkomst. Op die manier wordt veel genetische diversiteit in kaart gebracht. Dan spreekt men niet meer van een menselijk referentiegenoom, maar een menselijk pangenoom: een verzameling van heel wat genomen die samen wél representatief zijn voor de menselijke diversiteit. Als volgende stap kunnen nieuwe genetische resultaten niet enkel vergeleken worden met één referentiegenoom, maar kan het volledige pangenoom gebruikt worden. Vanzelfsprekend zorgt dit wel dat er ineens nood is aan nieuwe softwareprogramma's, om de vergelijking van sequencing data met al die genomen mogelijk te maken en daar nog eens medisch zinvolle conclusies uit te trekken. Dat is het terrein van de bioinformatica, en deze innovaties gaan wellicht in de vorm van zogenaamde graph genomes zijn. Hierbij wordt gewerkt met een kluwen van verschillende parallel lopende referentie genomen, waarbij ruimte voorzien is voor kleine en grote genetische diversiteit. Voor nieuwe genetische resultaten kan dan gezocht worden naar welke route in deze kluwen het beste overeenkomt met dit individu.
Het menselijke DNA heeft nog lang niet al haar geheimen prijsgegeven, en voor heel veel genetische aandoeningen is er nog veel te ontdekken. Maar met de verbeteringen van ons referentiegenoom, nauwkeurigere en langere sequencing technologieën en krachtige bioinformatica algoritmen wordt het de komende jaren nog erg interessant.