De Nobelprijs voor Scheikunde gaat dit jaar naar een celbioloog, een fysicus en een computerwetenschapper. David Baker slaagde erin om compleet nieuwe eiwitten te maken met Rosetta. Demis Hassabis en John M. Jumper gebruiken de kunstmatige intelligentie van AlphaFold2 om de structuur van zowat alle bekende eiwitten te voorspellen.
Zonder eiwitten geen leven. Eiwitten initiëren en sturen alle chemische reacties die de basis van het leven vormen. Ze doen dienst als hormonen, signaalstoffen, antilichamen en de bouwstenen van verschillende weefsels. Eiwitten bestaan meestal uit een twintigtal verschillende aminozuren.
De aminozuren zitten aan elkaar in lange slierten. Die vouwen zich op tot een driedimensionale structuur die de functie van het eiwit bepaalt. Sinds de jaren 1970 probeerden onderzoekers eiwitstructuren te voorspellen op basis van aminozuursequenties, maar dat was erg moeilijk. Vier jaar geleden kwam er echter een doorbraak.
Voorheen duurde het vaak jaren om een eiwitstructuur te verkrijgen. Nu kan het in een paar minuten
In 2020 presenteerden Demis Hassabis en John Jumper het AI-model AlphaFold2. Daarmee konden ze de structuur voorspellen van zowat alle tweehonderd miljoen eiwitten die wetenschappers tot nu toe hebben ontdekt en beschreven. Sinds hun doorbraak is AlphaFold2 gebruikt door meer dan twee miljoen mensen uit 190 landen. Heel veel wetenschappers gebruiken AlphaFold2, zoals om antibioticaresistantie beter te begrijpen of enzymen te analyseren die plastic kunnen afbreken.
In 2003 slaagde David Baker er met hulp van Rosetta in om een nieuw eiwit te ontwerpen uit aminozuren. Sindsdien heeft zijn onderzoeksgroep het ene na de andere eiwit geproduceerd, die werken als geneesmiddelen, vaccins, nanomaterialen en piepkleine sensoren.
De beste eiwitontsleutelaar wint
Als scheikundigen de aminozuurvolgorde van een eiwit kennen, zouden ze de driedimensionale structuur van het eiwit moeten kunnen voorspellen. Dat was vijtig jaar lang de grote uitdaging van de biochemie. Om de kwestie een beetje vooruit te laten gaan, startten onderzoekers in 1994 Critical Assessment of Protein Structure Prediction (CASP), dat uitgroeide tot een competitie. Onderzoekers van over de hele wereld namen de uitdaging aan en trachtten eiwitstructuren te voorspellen op basis van aminozuursequenties.
De overeenkomst tussen de voorspellingen van de wetenschappers in de competitie en de werkelijke structuren was jarenlang hoogstens veertig procent. De grote doorbraak kwam pas in 2018, toen schaakgrootmeester, expert neurowetenschappen en pionier op het gebied van kunstmatige intelligentie Demis Hassabis zijn intrede deed. Hassabis is medeoprichter van DeepMind, een bedrijf dat AI-modellen ontwikkelde voor populaire bordspellen. In 2018 schreef hij zich in voor de dertiende CASP en bereikte met het model AlphaFold een score van bijna zestig procent, een spectaculaire verbetering.
Hassabis en zijn team wilden AlphaFold nog verbeteren, maar ze bleven steken op die zestig procent, tot John Jumper bij het bedrijf kwam werken. Die hield zich al jarenlang bezig met eenvoudige methoden om de dynamica van eiwitten te simuleren. In 2017 was hij net gepromoveerd toen hij geruchten hoorde dat Google DeepMind in het diepste geheim was begonnen met het voorspellen van eiwitstructuren. Hij stuurde hen een sollicitatiebrief.
Hij werd aangenomen, en Jumper en Hassabis leidden samen het werk dat het AI-model fundamenteel hervormde. De nieuwe AlphaFold2 maakte gebruik van transformers, innovatieve neurale netwerken die op een flexibelere manier patronen herkennen. Het team trainde AlphaFold2 op informatie van alle bekende eiwitstructuren en aminozuursequenties. Tijdens de CASP in 2020 werd duidelijk dat de vijftig jaar oude uitdaging van de biochemie voorbij was: AlphaFold2 presteerde meestal bijna even goed als röntgenkristallografie.
David Baker begon zich als student te verdiepen in celbiologie en raakte uiteindelijk gefascineerd door eiwitstructuren. In 1993 ging hij de grote uitdaging van de biochemie aan: met slimme experimenten begon hij te onderzoeken hoe eiwitten zich vouwen. Eind jaren negentig begon hij een programma te ontwikkelen dat eiwitstructuren kon voorspellen: Rosetta. Baker debuteerde in 1998 in de CAS met Rosetta, en in vergelijking met andere deelnemers deed hij het erg goed.
Nieuwe eiwitten vanuit het niets
Hij wilde nu Rosetta andersom gebruiken: een gewenste eiwitstructuur invoeren en een suggestie krijgen voor de aminozuursequentie. Zo zouden ze nieuwe eiwitten moeten kunnen maken vanaf nul. Nieuwe eiwitten werden tot dan toe gemaakt door bestaande natuurlijke eiwitten aan te passen, maar het aantal natuurlijke eiwitten is beperkt. Baker zei: ‘Als je een vliegtuig wil bouwen, begin je ook niet vanuit een vogel. Je leert eerst de beginselen van aerodynamica en je bouwt je vliegende machines op basis daarvan.’
De onderzoeksgroep tekende een eiwit met een nieuwe structuur en liet Rosetta vervolgens berekenen welke aminozuursequentie het gewenste eiwit zou hebben. Rosetta doorzocht daarvoor een database van alle bekende eiwitstructuren naar korte fragmenten van eiwitten met de gewenste structuur. Rosetta kon met haar fundamentele kennis van energie van eiwitten die fragmenten aan elkaar koppelen en een aminozuurvolgorde voorstellen.
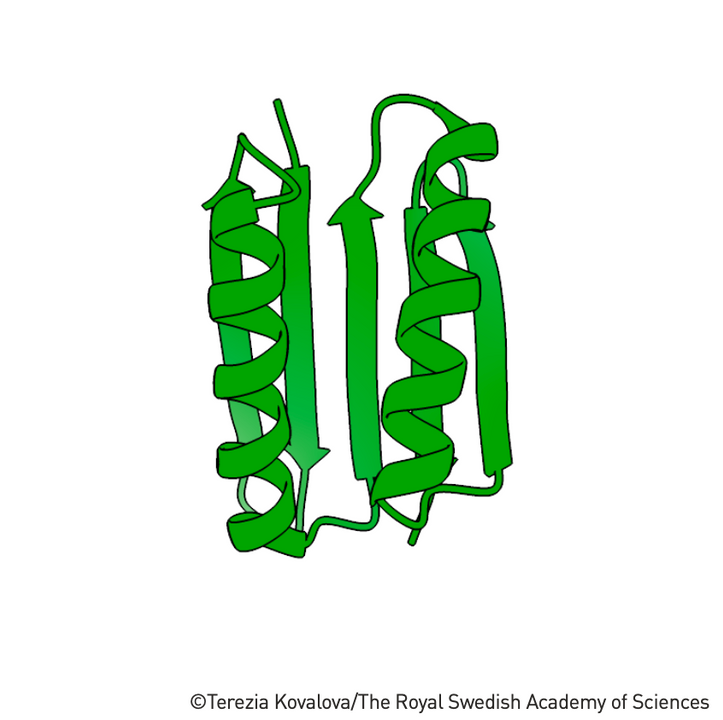
Om te onderzoeken hoe succesvol de software was, introduceerde Bakers onderzoeksgroep het gen voor de voorgestelde aminozuursequentie in bacteriën. Die produceerden het eiwit en volgens de röntgenkristallografie was de structuur de gewenste. Het bleek dus dat Rosetta echt eiwitten kon maken. Het nieuwe eiwit, Top7, had bijna precies de structuur die ze hadden ontworpen. Bovendien was het eiwit met zijn 93 aminozuren groter dan alles wat eerder was geproduceerd uit het niets. Baker gaf de code voor Rosetta vrij, zo kon de wereldwijde onderzoeksgemeenschap de software blijven ontwikkelen en nieuwe toepassingsgebieden ervoor vinden.
Toen Demis Hassabis en John Jumper hadden bevestigd dat AlphaFold2 echt werkte, berekenden ze de structuur van alle menselijke eiwitten. Daarna voorspelden ze de structuur van vrijwel alle tweehonderd miljoen eiwitten die wetenschappers tot nu toe hebben ontdekt. Google DeepMind heeft ook de code voor AlphaFold2 openbaar gemaakt, en iedereen kan er toegang toe krijgen.
AlphaFold2 is tot nu gebruikt door meer dan twee miljoen mensen uit 190 landen. Voorheen duurde het vaak jaren om een eiwitstructuur te verkrijgen. Nu kan het in een paar minuten. Na de CASP 2020 realiseerde David Baker zich het potentieel van AI modellen gebaseerd op transformers. Hij voegde er een toe aan Rosetta, en dat heeft ook het nieuwontwerp van eiwitten vergemakkelijkt.